Durante a minha Iniciação Científica, eu estudei muitos assuntos relacionados à Biologia e à Computação, mas descreverei a seguir somente os assuntos que acredito serem relevantes para este projeto.
Primeiramente, tive que estudar alguns conceitos básicos de Genética Molecular tais como ácidos nucléicos (DNA e RNA), códons, anti-códons, genes, genótipo, cromossomos, cariótipo e proteínas (em particular, aminoácidos). Estudei em detalhes a constituição dos DNAs e RNAs (nucleotíeos) bem como a estrutura dos mesmos (um ou dois filamentos de polinucleotídeos). Ao estudar as unidades básicas do ácidos nucléicos, os nucleotídeos, focalizei minha atenção nos constituintes destas unidades - fosfato, pentose (ribose ou desoxirribose) e base nitrogenada (adenina (A), timina (T), Citosina (C), Guanina (G) ou Uracila (U)), onde estas bases formam o que chamamos de "alfabeto genético" - e nas ligações entre elas (pontes de hidrogênio).
Ainda com relação à estes conceitos, estudei os mecanismos de duplicação celular, onde ocorre replicação do DNA, e de produção de proteínas, para as mais diversas finalidades específicas, mas com o propósito geral de controlar a célula.
Com relação à síntese de proteínas, alguns pontos importantes estudados foram os tipos de RNAs envolvidos (RNAm - RNA mensageiro, RNAt - RNA transportador e RNAr - RNA ribossômico), as relações entre eles, a relação entre eles e os DNAs, a relação entre os RNAs transportadores e os aminoácidos transportados por eles (que são 20 tipos diferentes), os genes expressos, os tipos de genes (estrutural, regulador, promotor e operador) e a interferência dessas proteínas em outras sínteses e outras reações, formando redes de regulação gênica.
Depois destes conceitos, comecei a estudar algumas técnicas de mapeamento e seqüenciamento genético, onde posso destacar a técnica de seqüenciamento usando amplificação de DNA através de clonagem e PCR (Polymerase Chain Reaction).
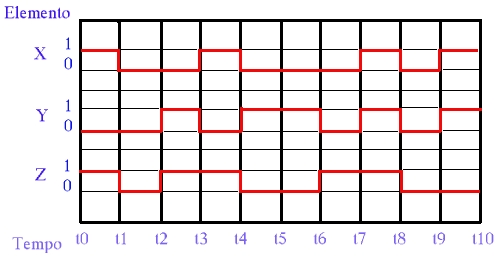
Para entender melhor os processos biológicos que regem o funcionamento de células, fiz uma pesquisa intensa em papers e na Internet sobre redes de regulação gênica ("Biological Pathways"), estudando alguns modelos de redes que descrevem os princípios que governam as interaçõs entre genes e proteínas. Dentre os modelos estudados, o escolhido para o projeto foi o booleano (figura 2), pois exibe propriedades dinâmicas semelhantes às de sistemas vivos, tal como auto-organização. Neste modelo, os genes e proteínas são considerados como elementos binários (expressos/não-expressos, 1/0) que interagem entre si até um estado final. Durante as interações, temos acesso aos estados dos elementos, sabemos quais elementos estimulam ou inibem outros elementos. Com isso, pode-se inferir a estrutura geral de uma rede em particular.
 |
| Figura 2: Gráfico de Simulação de Rede Booleana. |
No gráfico acima, podemos ver um exemplo do modelo booleano. Nele, X , Y e Z são elementos de uma rede e as curvas mostram quando cada um deles está ativo (estado 1) ou inativo (estado 0), em função do tempo.
O estudo desses assuntos foi completado com um tema cuja impotância aumenta a cada dia, que é a comparação de seqüências genéticas em bancos de dados. A busca de sequências em bancos de dados permite determinar quais das centenas de milhares de sequências presentes no banco podem estar relacionadas a uma dada sequência. Nesse tipo de ambiente, a operação básica consiste em alinhar uma sequência de consulta com as sequências do banco de dados. Os resultados são apresentados como uma lista ordenada de "hits" , seguidos por uma série de alinhamentos individuais, além de várias pontuações e estatísticas.
Os atuais bancos de dados de sequências já são gigantescos, e continuam a crescer numa taxa exponencial. Isso torna a aplicação de programação dinâmica pura inviável, obrigando o uso de heurísticas, que aumentam bastante a velocidade dos alinhamentos (mas com uma pequena probabilidade de perder alinhamentos verdadeiros).
Os dois programas de busca de sequências mais usados na atualidade são BLAST e o FASTA . Além destes dois, eu fiz buscar no PFAM , PSORT e COG .
Para representar as redes, era preciso escolher uma estrutura de dados adequada. Em virtude disso, a estrutura de grafos foi escolhida para tal propósito. Com isso, tornou-se essencial o conhecimento de algoritmos para desenhar grafos de forma que a relação entre os elementos do grafo seja facilmente entendida somente olhando o desenho. Fiz uma pesquisa extensa na Internet sobre o assunto e estudei vários algoritmos de desenho de grafos (forma de ávore, forma planar de grafo não dirigido, forma planar de grafo dirigido, entre outras). Tive que relembrar alguns algoritmos clássicos de grafos, tais como busca em largura, busca em profundidade (usado para desenhar grafos em forma de árvore), busca por ciclos (usado em grafos com forma circular), algoritmo das distâncias, entre outros.
|
|
Na figura 3, podemos notar que o grafo desenhado possui muitos cruzamentos entre as arestas, enquanto que na figura 4 o grafo não possui cruzamentos. O desenho da figura 4 é obtido após a aplicação de um algoritmo de planarização. Como pode-se ver, neste caso o critério utilizado para desenhar o grafo na figura 4 é o número de cruzamentos, que deve ser mínimo.
| << Anterior | | | Índice | | | Próximo >> |